MEGAを使って系統樹を描いてみました

この6月、一昨年からのずっと気になっていたキノコに再び巡り合うことが出来た。
さっそく阿部さんのところに送ってDNAを調べてもらったら非常に興味深い結果が出てきた。
Entoloma nipponicum(フチドリモミウラタケ) と99.3%の一致。
同じ日、1kmほど離れたところで見つけた「これはどう見てもフチドリモミウラタケだろう」と思われたキノコ。それも阿部さんに調べてもらったところ、そちらはフチドリモミウラタケと100%一致していた。
この「0.7%の違い」とはなんだろうか・・・?
僕の中では答えが出るはずもないのだが、少なくとも傘の色の違いやヒダの縁取りの色の違いなど細かい違いがあった。
0.7%の違い、というのはそれらの「色の違い」を産み出すのだろうか?なんて考えていた。
すると別の日、まったく別の場所で見つけたEntoloma(Cyanula亜属)をまた阿部さんに調べてもらったところ驚くべき結果が出たのだ。

Entoloma nipponicum(フチドリモミウラタケ) と99.3%の一致!!。
どういうことなのか・・・。
こちらはフチドリモミウラタケとは全然違う色ではないか!!!
しかし、傘の形状、鱗片、柄の細鱗片やヒダの縁取りなど色以外では共通する部分がめちゃくちゃ多い。
ちなみに、その場所から300mぐらい離れた場所で「これはどう見てもフチドリモミウラタケ」というものを発見したのですが、そいつのDNAはまだ取れていないのですが、今回の結果があまりにも衝撃的だったので、どうなっているのか?と思って系統樹を作ってみました。
系統樹はまだまだ「なんちゃって系統樹」のレベルで、設定値などは教えられたものをそのまま使っており、そのものを理解しているわけではない、ということを前提に、あくまでも「こんな風に系統樹を作っていくんだなぁ、、」という感じで読んでもらえればと考えております。
またこの系統樹作成についても阿部さんから沢山教えてもらったので、改めてここでお礼を申し上げておきます <m(__)m>
系統樹作成ソフト「MEGA Ⅹ」
まず系統樹を作成するにあたっては「MEGA Ⅹ」というソフトを使って行っています。
ここではインストール方法やMEGAの詳しい使い方などは書きませんので(書けないという方が正確ですね)、もしインストールして試してみたいという人がいれば、他にその様なサイトがありますので、そちらを参照してください。
ここではあくまでも僕の「使い方メモ」程度に手順をまとめていきますので、ご了承ください。
またもし「こうした方がいいよ」というのがあればぜひぜひご指摘下さいませ。
MEGAのダウンロードサイト(現在はMEGA 11)
https://www.megasoftware.net/
参考にしたサイト
「系統樹の書き方(MEGAを使用した系統樹の書き方)」
https://bio-and.info/post/DNA_Analysis/PhylogeneticTreeConstruct.html
1.Fastaファイルから類似の種を検索する
DNAのシーケンスデータとして「Fasta形式」のデータをいつも阿部さんから送って頂きます。
「Fasta形式」のデータというのはこういうものです。
>2023-06-09_230608_ABE_ITS_HWK9_E10(フチドリモミウラタケ))
AAGATTGATAAATTATAAGCAAGATCTTTTCTTTTGAAAAACACTTGTTCATTAGTGATATAGATAATTATCATATCAAA
GATGATTCAAGTGATTAATCTTGCTAATGCATTTAAGAAGAGCCGAGTCGCGAAGAACCAGCAAGCTTCCAAAGTCCAAG
CCTAACTAAGCAAAACAGCTTTGAAAGGTTGAGAATTTCATGACACTCAAACAGGCATGCTCCTCGGAGTACCAAGGAGC
GCAAGGTGCGTTCAAAGATTCGATGATTCACTGAATTCTGCAATTCACATTACTTATCGCATTTCGCTGCGTTCGTCATC
GATGCAAGAGCCAAGAGATCCGTTGTTGAAAGTTGTATTAAATTTTTGTTTATAACCATTCTAATACTTTTAATTGGAGT
ATATGTTTGAAACATAGATCTGAAAAGATACAAGAAAAAGCTGGCATAAAAAACCACAGCAATTCCTCTGACCAAGACAT
TAAAATCTTGAAAAAA
なんだか不思議な文字列が並んでおりますが・・・・
「これなんだろう?」
「どうやって使ったらえぇのじゃ?」
と最初思っていたのですが、この文字を見ていくと「A」「G」「T」「C」という4つの文字のみが使われていて、それぞれ
G:グアニン
C:シトシン
A:アデニン
T:チミン
という核酸塩基の配列となっているのです。
この配列はその種を特定する重要な要素となっており、この配列を他の種のものと比較(Blast)することによって、その種の「位置」が分かるのですね。
Blastとは?
「BLAST」(Wikipedia)
Blastとは 「Basic Local Alignment Search Tool)」の略で、バイオインフォマティクスでDNAの塩基配列あるいはタンパク質のアミノ酸配列のシーケンスアライメントを行うためのアルゴリズムをいい、またそのアルゴリズムを実装したプログラムをいう。
https://ja.wikipedia.org/wiki/BLAST#:~:text=BLAST%20(Basic%20Local%20Alignment%20Search,%E5%AE%9F%E8%A3%85%E3%81%97%E3%81%9F%E3%83%97%E3%83%AD%E3%82%B0%E3%83%A9%E3%83%A0%E3%82%92%E3%81%84%E3%81%86%E3%80%82
またBlastを行えるサイトとしては以下のサイトが有名です。
「National Library Of Medicine」
https://blast.ncbi.nlm.nih.gov/Blast.cgi
ではここのサイトで上記フチドリモミウラタケのDNA配列を読み込ませてみましょう。
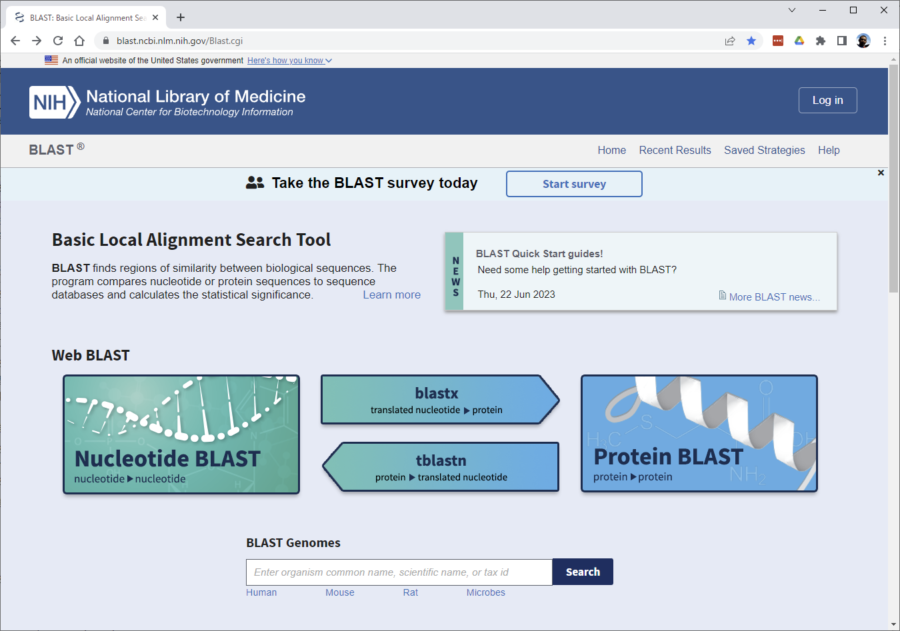
このページの「Nucleotide BLAST」というボタンをクリックしましょう。
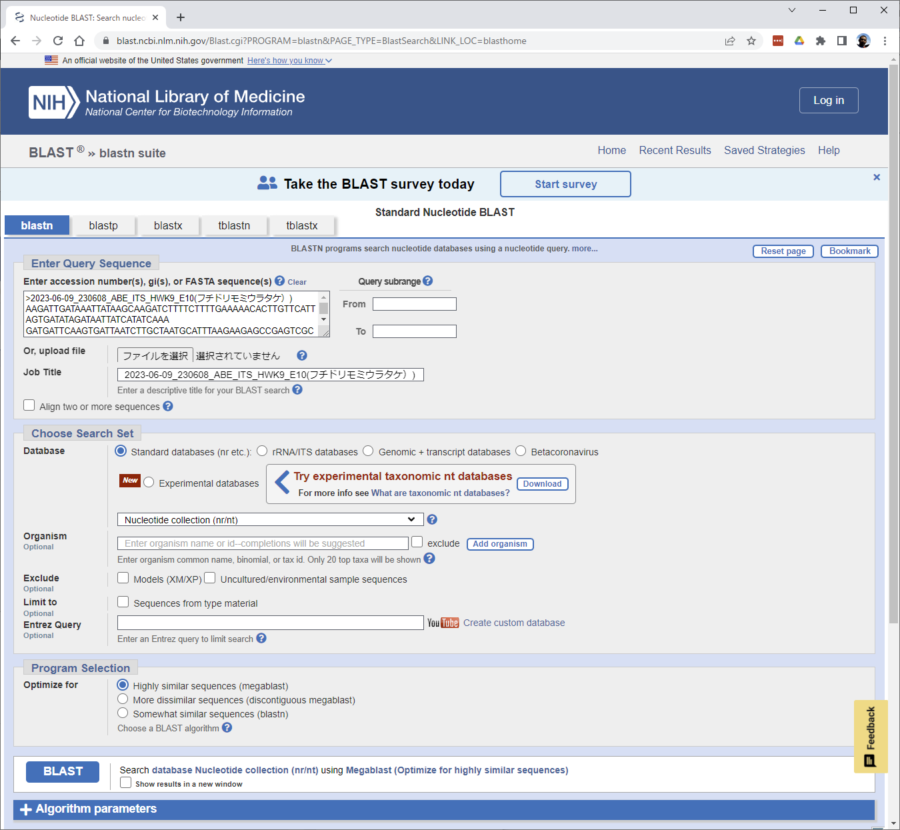
「Enter accession number(s), gi(s), or FASTA sequence(s) 」と書かれているBOXの中に、フチドリモミウラタケのDNA配列をコピペします。
その後、左下の「BLAST」ボタンをクリックしてください。
するとしばらくして以下の結果が表示されます。
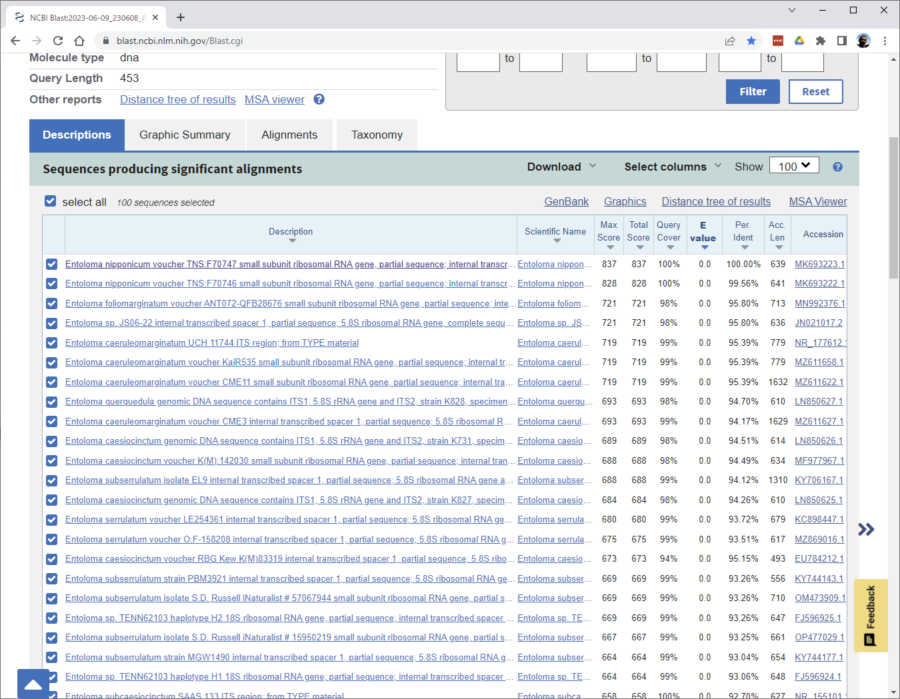
この中で注目なのは表のタイトル「Per Ident」という項目です。
今回のDNA配列は既に登録してある「Entoloma nipponicum voucher TNS:F70747」のものと100%一致している。
ということを示しています。
2.FASTAデータを取得する
では先ほどの表の一番右にある「Accession」というタイトルのところにある番号をクリックしてみましょう。
今回のものは「MK693223.1」というものです。
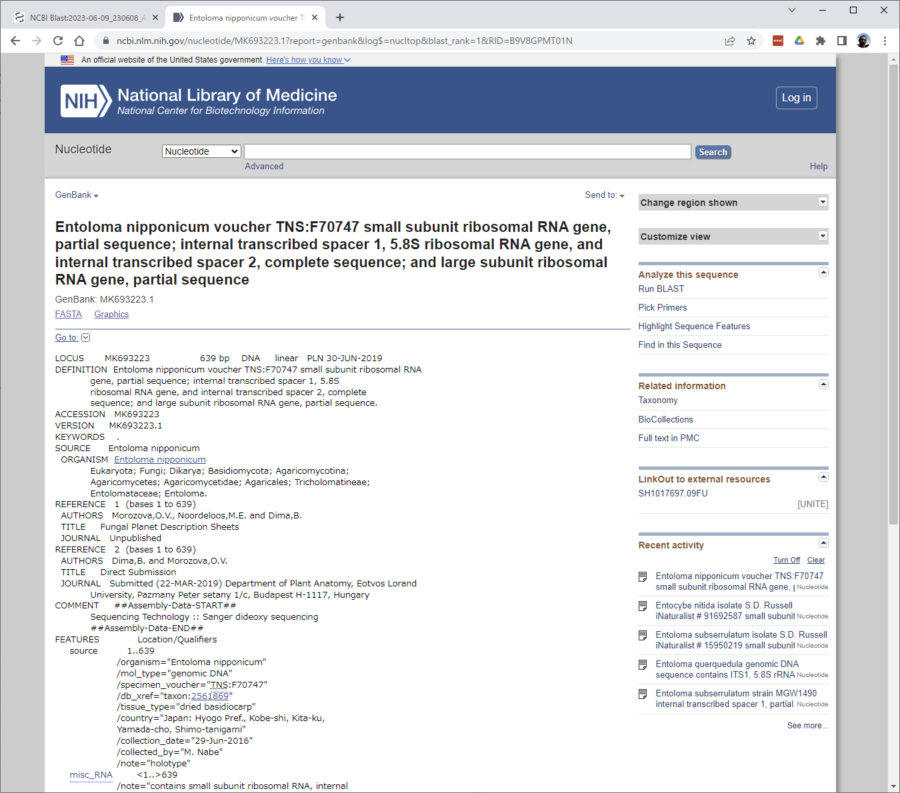
これを見ると登録した人の名前や採取地、採取した人の名前などが記されているのが分かります。
で、このページの「FASTA」というリンクをクリックしてみましょう。
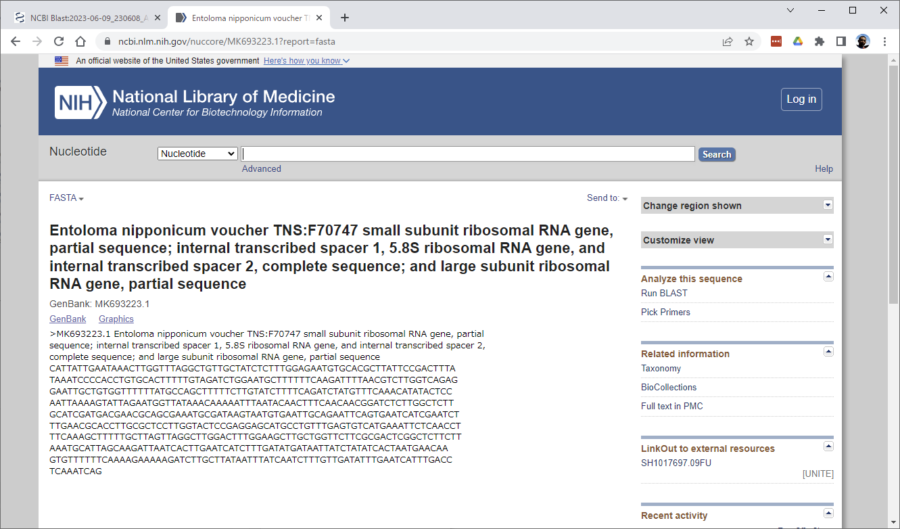
FASTA形式のシーケンスデータが表示されました。
これをコピーしてFASTAファイルにペーストしていきます。
※これを「Per Ident」が高いものから順番に20個ぐらいコピペして一つのファイルに入れていきましょう。
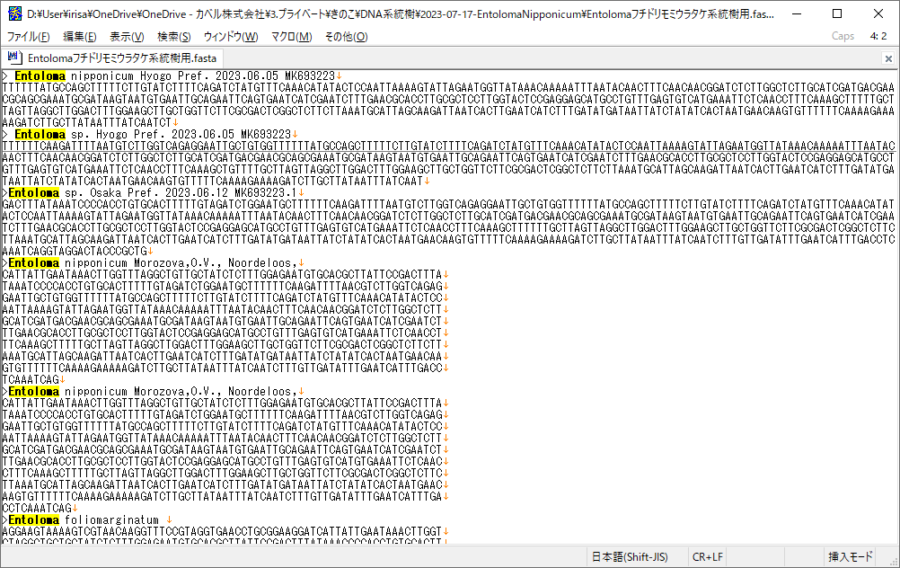
そして最後に「OUTGROUP(外群)」となる種のFASTAを取得しましょう。
ここでは88.1%の一致率である「Entoloma exile」という種のFASTAを」取得し最後に追加します。
OUTGROUPとは?
http://stat.sys.i.kyoto-u.ac.jp/titech/class/enshu200309suitei.html
outgroupは系統樹の根を指定している。
DNAシーケンスを比較して得られる系統樹は本来根の位置が分からない木(unrooted tree)である。そこであらかじめ最も遠縁と分かっている生物(すなわちアウトグループ)を含めて系統樹を推定し,このアウトグループのついている枝が根の位置であると考える。
—
3.系統樹を作成する準備
それでは系統樹を作成する準備を始めます。
まずはMEGAを起動します。
ボタンがずらっと並んでいるのですが、この中の「ALIGN」という左端のボタンをクリックするとメニューが開き、その中から
「Edit/Build Alignment」というメニューをクリックしますと以下の様なダイアログが開きます。
「Retrieve a sequence from a file」を選んだまま「OK」ボタンを押します。
先ほど編集したファイルを選択し、「開く」ボタンをクリックします。
すると、FASTAデータの内容がこんな感じで表示されます。
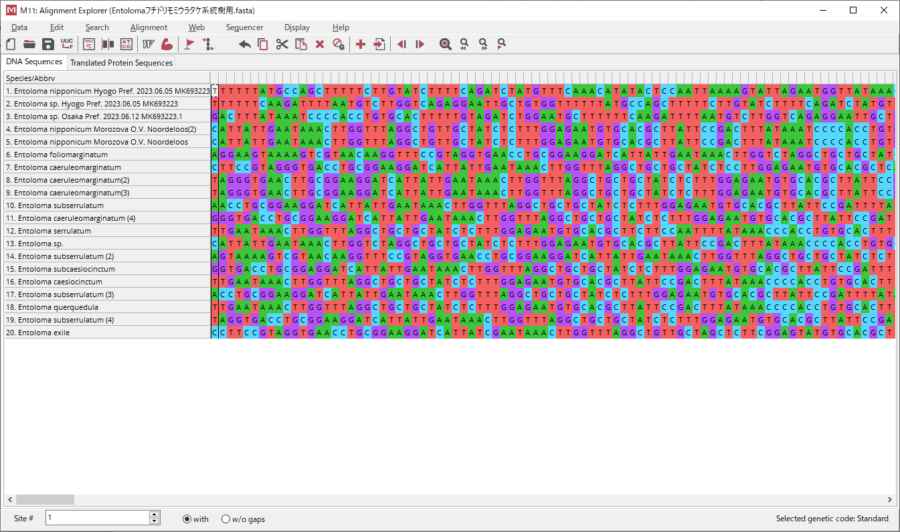
ここでずらっと並んでいるアイコンから「筋肉」のアイコン(左から9個目)をクリックし、「Align DNA」を選択してください。
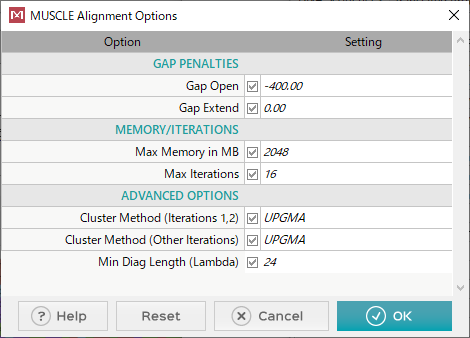
上記画面に対して「OK」ボタンを押すと以下の様な画面になります。
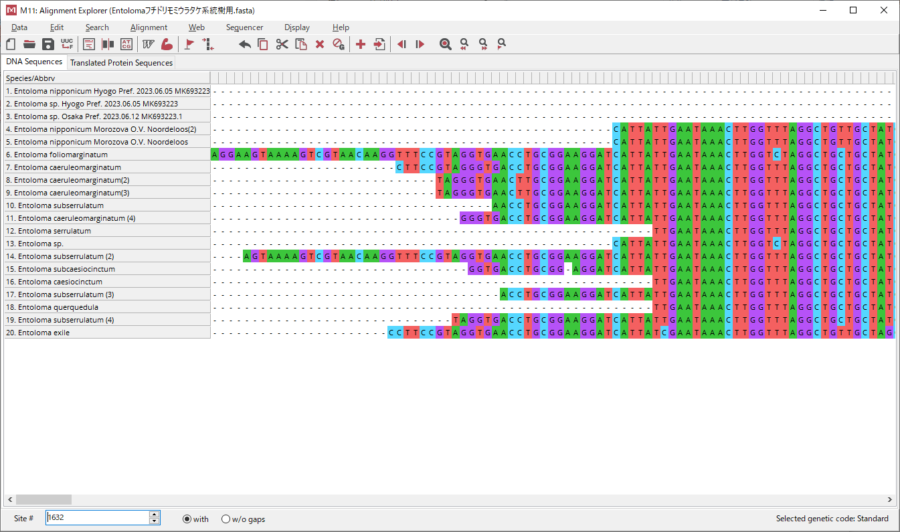
これがアライメントされた結果となります。
アライメントとは?
「微生物の系統樹,どう描くの?」
アライメントは比較する塩基(アミノ酸)配列の各座位の比較対象を決めるための位置合わせ,あるいはその結果として複数の塩基配列が整列配列されたものを示す。祖先を共有する二つの遺伝子配列同士を比較する場合,一方の塩基配列の座位に挿入や欠損が起きた場合にはどちらかの配列にギャップを入れて,他の座位が比較できるように位置合わせをする必要がある。
https://www.sbj.or.jp/wp-content/uploads/file/sbj/9110/9110_yomoyama.pdf
この後に、「DATA」メニューから「Phylogenetic Analysis」をクリックすると元の画面にアイコンが作成されます。
4.系統樹の作成
元の画面のアイコンの一つ「TA」と書いてあるアイコンをクリックすると、「Sequence Data Exploler」が起動します。
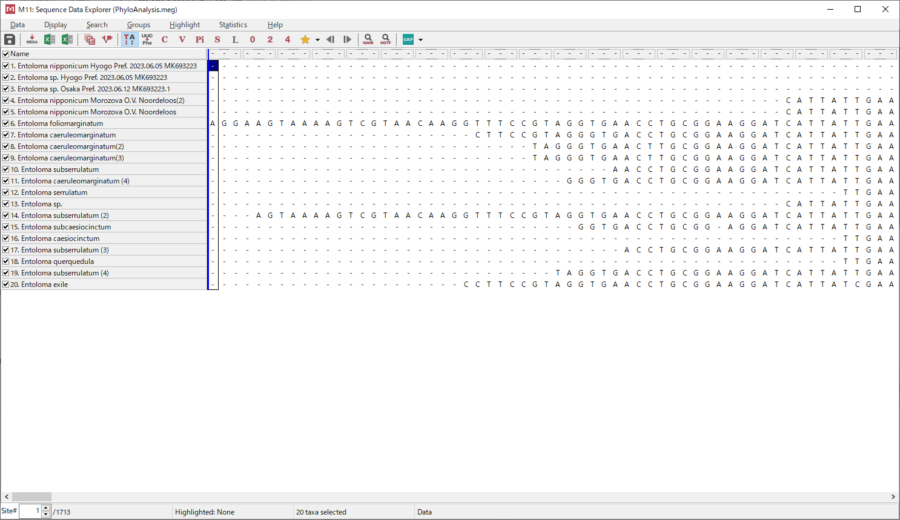
この画面から「Data」をクリックし、「Select & Edit Texa/& Groups…」をクリックします。
ると以下の様な画面が開きます。
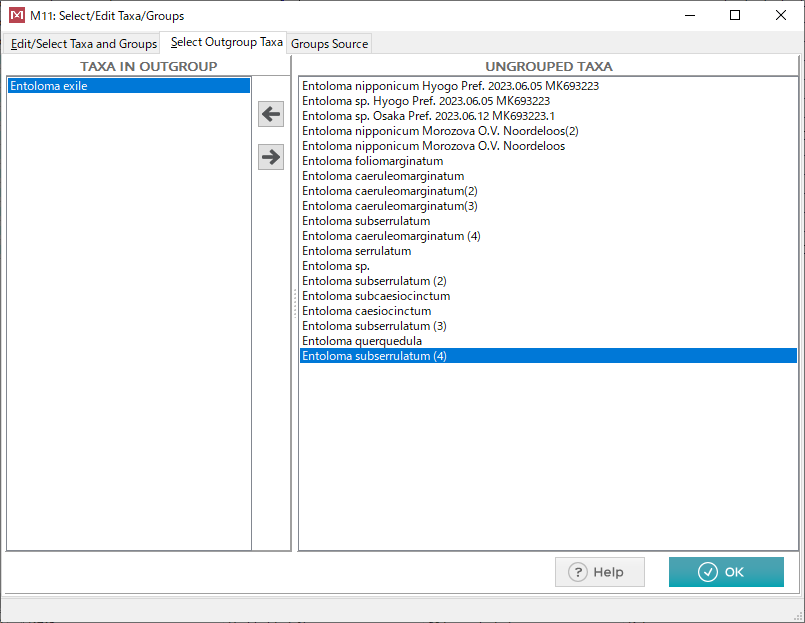
ここで「Select Outgroup Texa」というタブを開いて一番下に追加した「Entoloma exile」をクリックし「←」ボタンでOUTGROUPに移動し、「OK」ボタンを押してください。
これで、「Entoloma exile」をOUTGROUPに設定したことになります。
それではメインの画面に再び戻ります。
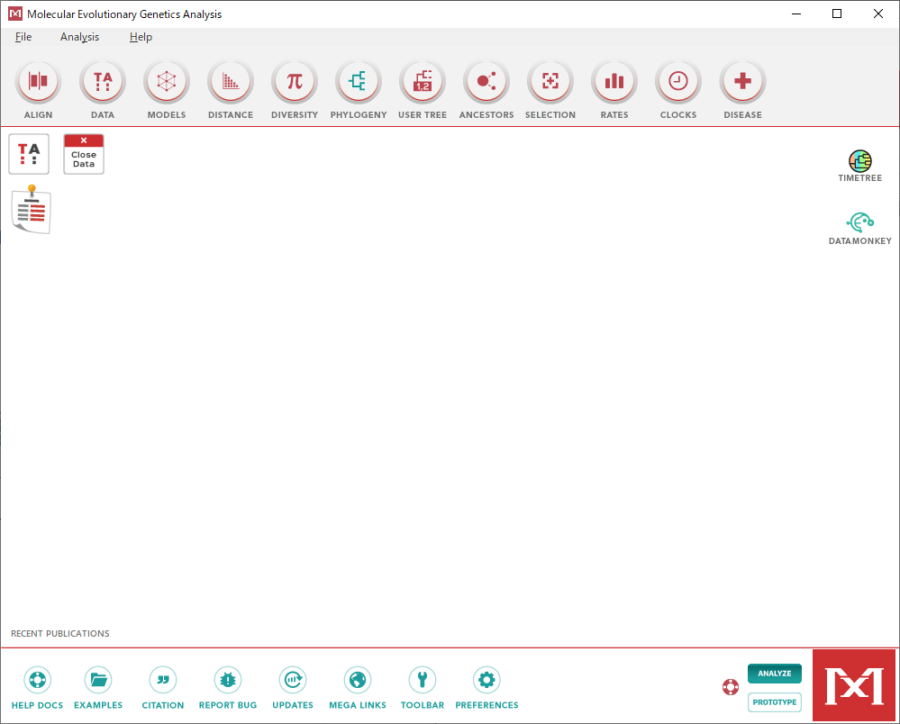
左から6番目のボタン「PHYLOGENY」を押して、「Construct/Test Maximum Likelihood Tree…」をクリックします。
※一般的には「Likelihood Tree」が良く使われているそうです。
すると以下の様なダイアログが開きます。
ここのパラメタに関しては詳しいことは分かりません (#^.^#)
※阿部さんのお勧めパラメタに設定しております。
すると以下の様な進捗画面が表示され、しばらくすると系統樹が表示されます。
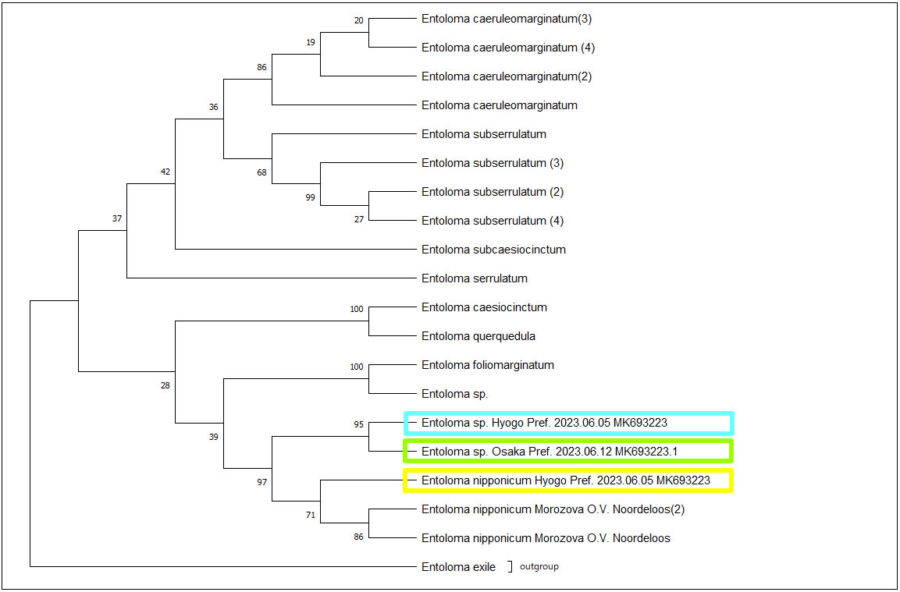
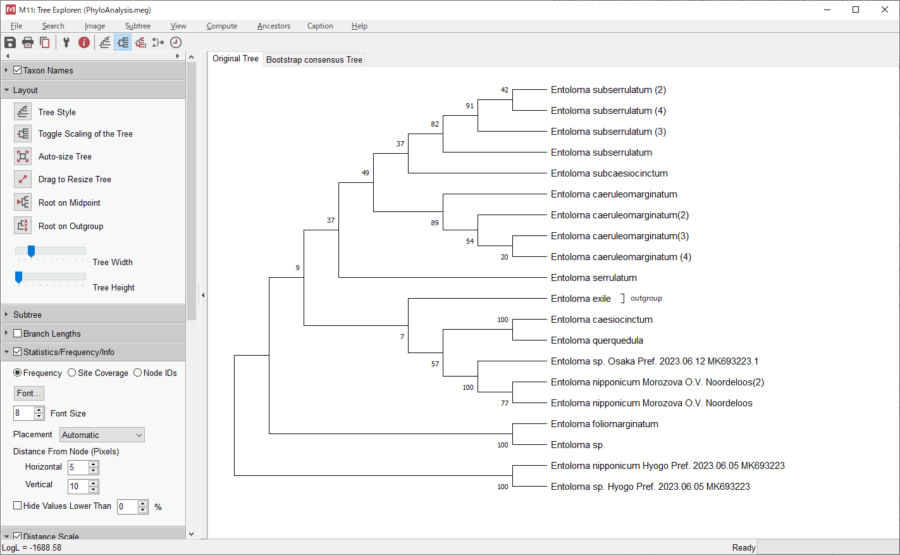
作成された系統樹。
色を塗っているのは今回DNAのデータを送ってもらったもの。
これで見ると位置関係が良くわかりますね。
5.追記:系統樹がおかしい時は?
上記で作成された系統樹は以下の点で正しく作成されていると思われます。
- Entoloma nipponicum Hyogo Pref. 2023.06.05 MK693223はEntoloma nipponicum と同じクレードにある
- Entoloma sp. Hyogo Pref. 2023.06.05 MK693223は少し離れていて
- Entoloma sp. Osaka Pref. 2023.06.12 MK693223.1も少し離れている
- 上記2つ共にEntoloma nipponicum Hyogo Pref. 2023.06.05 MK693223の近くにいる
- Entoloma exileは正しく OUTGROUP の位置にある
しかし、この様に綺麗な系統樹が描けない場合があった。
その結果がこれです。
分かりますか?
これは以下の2つのDNA配列がおかしいことを示しています。
- Entoloma nipponicum Hyogo Pref. 2023.06.05 MK693223
- Entoloma sp. Hyogo Pref. 2023.06.05 MK693223
これでかなり悩みましたが、別件で同じようなことがあったので阿部さんに相談したところ
「配列が逆向きじゃないか?」
との指摘を受けたことがあったので、上記2つの配列を以下のサイトで逆にしてみました。
「DNA逆向き相補鎖」
https://www.vectorbuilder.jp/tool/dna-reverse-complement.html
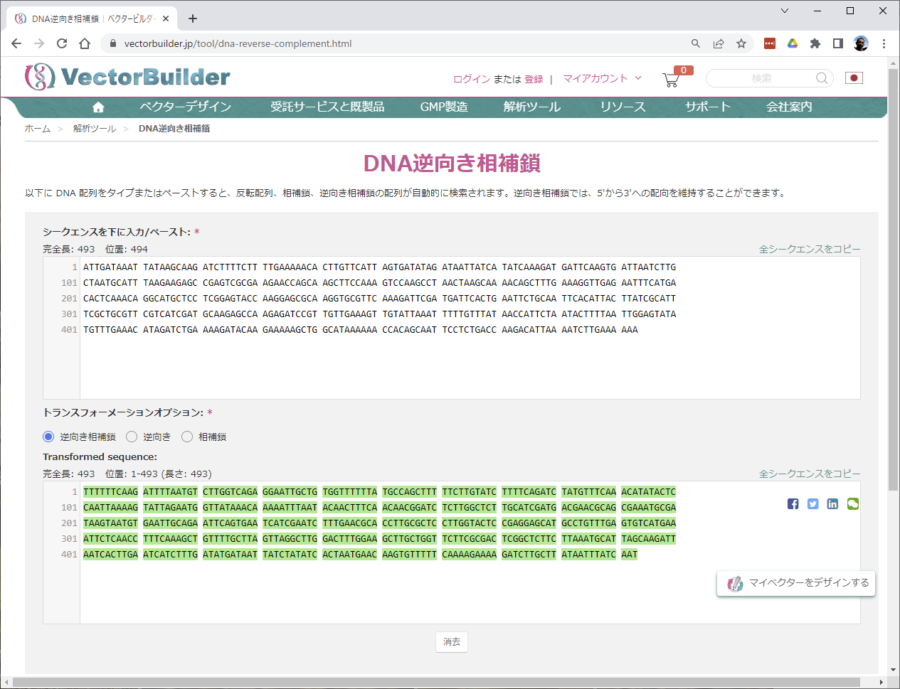
上に元のシーケンスデータをコピペするだけで、下のエリアに逆向きになったデータが表示されるという素敵なサイトです (#^.^#)
このデータをFASTAファイルに貼り付けなおして再度系統樹を作成すると以下の様な正しい系統樹が作成されます。



